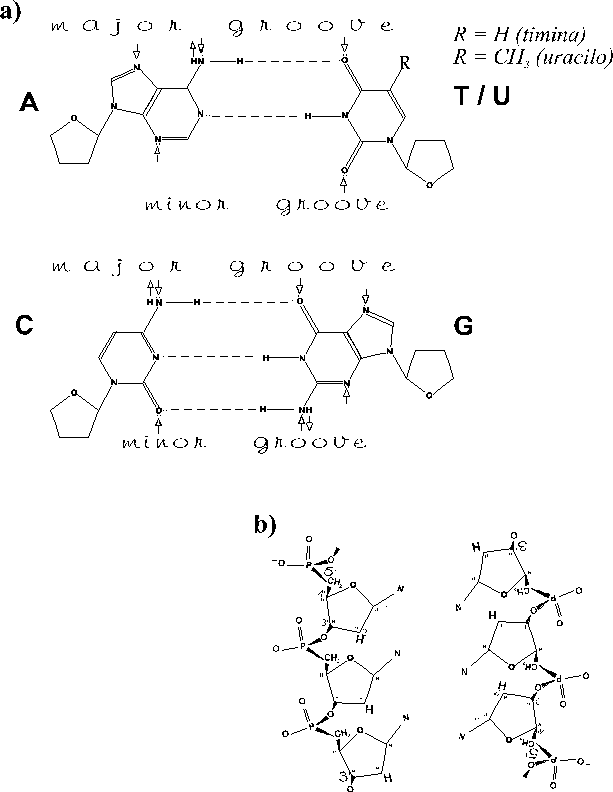
A molécula de DNA (figura 9) presente nos cromossomas tanto das células procarióticas como eucarióticas é constituída por duas cadeias poliméricas de resíduos de desoxirribonucleótidos. Duas componentes se complementam na estrutura do polímero: por um lado um "esqueleto" covalente para cada cadeia (figura 9b), mantido pelas ligações éster entre o carbono 3' de cada desoxirribose e o fósforo α do resíduo seguinte na mesma cadeia (ligações 3'–5'); e a "escadaria" de pares purina-pirimidina (figura 9a) que define a complementaridade entre as duas cadeias e também a sua orientação anti-paralela mantida por pontes de hidrogénio (não-covalentes).
Cada cadeia é polimerizada por condensação entre trifosfatos de desoxirribonucleótidos (dNTPs) e a extremidade 3'-OH de uma cadeia pré-existente, com a libertação de pirofosfato. Daí que cada cadeia linear de DNA tenha as extremidades livres não esterificadas, uma delas contendo um grupo trifosfato intacto, na posição 5' da primeira desoxirribose, e a outra contendo um grupo álcool 3' da última desoxirribose livre. Deste modo se diz que cada cadeia tem uma orientação 5' → 3'. As moléculas de DNA circulares (cromossomas de células procarióticas, plastos, mitocôndrias, certos vírus, plasmídeos, etc.) estão completamente esterificadas.
Dada a posição dos grupos fosfato no polímero, a sua carga negativa (a pH neutro) torna a molécula altamente hidrofílica. Em consequência disto, as moléculas que interagem com o material genético (nomeadamente proteínas) têm em geral cargas positivas necessárias a estabelecerem-se ligações iónicas com o DNA.
A desoxirribose do DNA difere da ribose do RNA pela substituição do grupo álcool 2' por um átomo de hidrogénio, que se sabe conferir ao DNA uma grande estabilidade termodinâmica, importante para a preservação da integridade dos cromossomas a todos os níveis da estrutura (o RNA é quimicamente muito mais reactivo que o DNA).
Os pares purina-pirimidina são perfeitamente planos, e através das interacções hidrófobas entre pares sucessivos promovem o enrolamento helicoidal nas moléculas de cadeia dupla. As regras de complementaridade entre as duas cadeias do DNA são as seguintes: cada nucleótido de purina (adenina ou guanina; símbolo genérico R) emparelha com uma pirimidina (timina ou citosina; símbolo genérico Y), as adeninas com as timinas (pares A::T, estabilizados por duas pontes de hidrogénio), as guaninas com as citosinas (pares G::C, estabilizados por três pontes de hidrogénio); no RNA, não ocorrem timinas, mas uracilos (U) que emparelham também com as adeninas por duas pontes de hidrogénio. Os pares G::C são, por terem mais uma ponte de hidrogénio, termodinamicamente mais estáveis que os A::T/U, o que tem diversas implicações não só para a dinâmica de interacção do DNA com as proteínas como em aspectos práticos da manipulação in vitro do DNA e do RNA.
Este modelo molecular (forma B), proposto por Watson e Crick, foi adoptado não só porque explica os dados analíticos elucidativos da estrutura do DNA, mas também porque está conforme a todos os dados resultantes do estudo da duplicação cromossómica. Outros modelos (formas A e Z) são aplicáveis a certos segmentos do material genético, podendo estar-lhes associadas certas implicações funcionais.