Figura 10 — Duas replicações sucessivas na ausência de nucleótidos
radioactivos e a consequente redução para metade da concentração de
radioactividade por cromátide. S e M são fases do ciclo celular. |
A replicação do DNA é semi-conservadora, isto é, o material pré-existente vai continuar intacto findo o processo, metade em cada uma das cromátides resultantes (figura 10). As pontes de hidrogénio que mantêm a dupla cadeia desnaturam-se para que cada uma das cadeias simples possa servir de "forma" ou molde (template em inglês) ao longo da qual vai ser sintetizada, no sentido 5' → 3', uma cadeia complementar.
A replicação do DNA envolve várias actividades enzimáticas:
i) a polimerase do DNA propriamente dita, que reconhece em cada posição o nucleótido da cadeia-molde, selecciona o dNTP que lhe deve ser complementar, e cataliza a condensação com a cadeia complementar já formada;
ii) a exonuclease 3', que rectifica eventuais erros de emparelhamento imediatamente após a integração de um novo resíduo nucleotídico, permitindo a integração do nucleótido correcto;
iii) diversas helicases, necessárias à desnaturação da cadeia dupla (que perde o seu carácter helicoidal, daí o nome que lhes é atribuído);
iv) a primase, que inicia a síntese das novas cadeias complementares em vários pontos da cadeia-molde;
v) a exonuclease 5', que promove a continuidade entre os fragmentos sintetizados, re-sintetizando-os, contribuindo também para a correcção de erros de emparelhamento.
Estas actividades são complementadas por ligases, que completam a continuidade das ligações éster das cadeias recém-sintetizadas, por topoisomerases necessárias ao alívio da tensão torcional resultante da abertura da dupla cadeia, e por sistemas de reparação suplementares, que ultimam a correcção de erros na síntese das novas cadeias, fazendo com que a probabilidade de se incorporarem mutações por erros de replicação atinja níveis muito baixos (que nos eucariotas se cifra na ordem dos 10–11 por par nucleotídico, em cada ciclo celular).
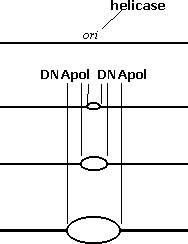 |
| Figura 11 — Formação e crescimento de uma bolha de replicação. DNApol indica a polimerase do DNA. |
As origens de replicação dos cromossomas eucarióticos são especificamente reconhecidas pela proteína que interage primeiro com elas, uma helicase. Esse reconhecimento faz-se através de grupos funcionais nos pares R::Y que ficam de um lado e doutro das duas cadeias polifosfato-desoxirribose, isto é, ao longo dos dois "sulcos" acessíveis (figura 9a). Cada par de Watson-Crick apresenta um padrão único de interacções possíveis nesses sulcos, de pontes de hidrogénio nomeadamente, e é da sucessão de pares nucleotídicos que resulta o padrão molecular especificamente reconhecido por proteínas como a helicase.
Por isso cada tipo de proteínas que interagem com o DNA deve "encaixar" com uma sequência (ou grupo de sequências) de pares nucleotídicos, específica para esse tipo; representam-se essas sequências de forma simplificada pelas letras dos nucleótidos (A, G, T, C) numa das cadeias do DNA apenas. É como se o DNA fosse um "texto" construído sobre um alfabeto destas quatro letras; por exemplo, para iniciar a "leitura" do DNA a ser replicado, há uma "palavra-chave" que só é reconhecida pelas helicases responsáveis pela formação de "bolhas" de replicação: todas as origens de replicação terão de conter uma "palavra-chave" apropriada, e só após estar realizado este reconhecimento molecular (e formada a bolha de replicação) o processo passa à leitura e cópia do restante "texto".
Os genomas eucariotas podem ser divididos em três classes de sequências, segundo a ordem de grandeza da sua repetitividade, isto é, do número de vezes que aparecem repetidas no genoma haplóide:
Repetitividade | Tipos de sequências |
|---|---|
105 – 106 cópias/ genoma | "satélites", retro-elementos |
102 – 104 cópias/ genoma | rDNA, tDNA, genes das histonas |
1 – 10 cópias/ genoma | restantes genes (genes com mRNA poli-A+) |
A proporção relativa das três classes varia bastante de genoma para genoma. A classe mais altamente repetitiva, embora pareça "não ter função" pois não contém genes, é de grande utilidade para o mapeamento cromossómico (cf. "mapas físicos", "QTLs"). A classe intermédia é formada pelos genes que são mais intensamente expressos nas células: os do RNA ribossomal (rRNA) e do RNA de transferência (tRNA) envolvidos na "maquinaria" de tradução dos diversos mRNA em todas as células, e os genes das histonas — que só se expressam quando na fase S a célula "interrompe" outras funções para a duplicação dos cromossomas.
As sequências menos repetitivas são precisamente aquelas que correspondem aos genes "específicos" de cada função, uns envolvidos em processos comuns a diversos tipos celulares (caso dos enzimas envolvidos na replicação do DNA), outros em processos restritos a certas células ou a certas circunstâncias (por exemplo na síntese de proteínas de reserva no endosperma das sementes, produção dos enzimas de bio-síntese da cutícula das folhas...).