A transcrição do DNA em RNA é o ponto de partida de toda a expressão fenotípica. Tal como na replicação, o acesso ao DNA é mediado pela interacção entre proteínas e certas regiões colocadas estrategicamente em relação ao segmento a ser transcrito; e também requer helicases que desnaturam a cadeia dupla para permitir-se a "leitura" da sequência de nucleótidos por polimerases. Mas as polimerases do RNA, enzimas que realizam a transcrição, utilizam apenas uma das cadeias do DNA como molde, permitindo que elas voltem a emparelhar no fim do processo; por isso o produto final da transcrição, ou transcrito primário, é uma molécula (de RNA) de cadeia simples. A polimerase de RNA termina a transcrição junto a sequências que indicam o final do mesmo gene que começou a ser transcrito, por isso a transcrição não é um processo que prossiga indefinidamente ao longo do cromossoma. Outra diferença em relação à replicação está na actividade de transcrição abranger toda a interfase, assim como o estado G0.
Mas nem todos os genes de um organismo são transcritos numa mesma célula. As células especializam-se em função do repertório de proteínas associadas a cada tipo de metabolismo, e também em função do doseamento de cada uma; por isso, na regulação da transcrição dos genes (quais os que são transcritos e quanto das respectivas proteínas é produzido por cada tipo de célula) está uma chave fundamental da diferenciação celular. Não deixa de haver proteínas com funções gerais (metabolismo energético, citosqueleto, polipéptidos ribossomais, histonas, etc.), codificadas nos genes "que mantêm a casa" (housekeeping), os quais são transcritos em todos os tipos de células; mas outras proteínas, que só se encontram em células ou em condições metabólicas bem determinadas, são codificadas por genes cuja transcrição sofre drásticas variações, segundo os casos podendo ser muito abundantes numas células e totalmente ausentes noutras, ou aumentarem de um nível "basal" de expressão para concentrações centenas de vezes (ou mais) superiores (e depois voltarem aos níveis basais), por exemplo em resposta a uma hormona (cf. secção seguinte).
A maquinaria enzimática da transcrição tem assim de "saber" responder a sinais muitíssimo diversificados e geralmente distintos dos que regulam a replicação. E ela própria se especializa, pois nos núcleos das células conhecem-se três tipos de polimerase do RNA: duas delas estão dedicadas à produção de moléculas de rRNA e tRNA, que são as mais abundantes e não são traduzidas; a polimerase III encarrega-se da síntese dos tRNA e rRNA 5S, enquanto a polimerase I da síntese dos restantes rRNA.
É assim que quase todos os genes (incluindo os housekeeping, os de metabolismos especializados, e também os dos vírus quando infectam uma célula-hospedeira, cf. "vírus e viróides"), são transcritos pela polimerase do tipo II. Por isso se tem dedicado a esta polimerase e à sua catálise a maior parte do esforço de pesquisa em transcrição, pois a sua actividade incide sobre toda a miríade de genes que codificam proteínas — onde também se vão encontrar praticamente todos os que são conhecidos pela análise mendeliana.
Nas mitocôndrias e plastos, lá se encontram as polimerases (de DNA e de RNA) análogas, codificadas nos respectivos genomas.
Regulação da transcrição pela polimerase II do RNAA transcrição de um gene não se realiza indiscriminadamente: há em princípio um tempo, um lugar e uma quantidade certas para a expressão desse gene, em ligação com dois aspectos muito importantes em Genética:
i) a diferenciação dos tipos citológicos presentes nos organismos pluricelulares: em cada tipo celular diferenciado há uma especialização cujo fundamento reside no repertório específico de loci que são expressos ou silenciados (isto é, no correspondente repertório de proteínas produzidas), o qual se define no percurso ontogénico que conduz a essa especialização; esse percurso, por sua vez, caracterizou-se pela sucessão de repertórios genéticos sucessivos, ou seja a diferenciação celular é a culminação de um programa genético de desenvolvimento somático;
ii) a resposta das células (ou, talvez mais propriamente, dos tecidos) aos estímulos recebidos, seja uma mudança da temperatura ou de outro parâmetro do meio, a presença de um corpo estranho, uma hormona, um vírus, etc.: um dado estímulo é reconhecido apenas por certo tipo de células-alvo equipadas para o traduzirem metabolicamente em sinais internos que levam à indução de loci até aí silenciados ou com uma expressão basal (isto é, não-induzida) muito baixa. Isto subentende haver nestas células, como corolário da sua diferenciação, uma expressão genética específica dos factores envolvidos nesses passos de reconhecimento, sinalização interna e indução.
Longe de ser suficiente para compreender a nível molecular o fundamento genético dos diferentes tipos de actividade celular e transições entre eles, designadamente as de crescimento (por exemplo meristemas e câmbios), de bio-síntese (praticamente todos os tipos celulares diferenciados, por exemplo glândulas, parênquimas clorofilinos, pelos radiculares, células pigmentares, etc., etc.) e apoptose (morte celular programada), a transcrição é o ponto de partida e como tal é alvo de intenso estudo nas mais diversas situações com relevância, por exemplo, para a Fisiologia.
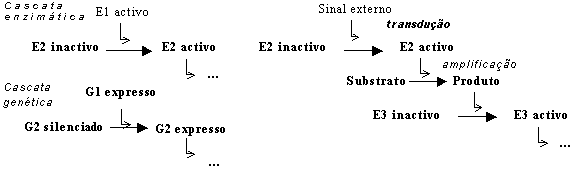
Os genes transcritos pela polimerase II do RNA podem ser genericamente classificados em dois tipos: genes estruturais e genes reguladores. As proteínas codificadas pelos genes estruturais, como o nome indica, integram a estrutura celular ou intervêm no metabolismo e transporte das diversas classes de biomoléculas que compõem as células. Os produtos dos genes reguladores são em geral muito menos abundantes, localizando-se conforme os casos no núcleo, no citosol ou na membrana celular para, directa ou indirectamente, regularem a expressão de outros genes. Esses, por sua vez, poderão ser estruturais ou reguladores de outros genes, neste caso indo regular outro conjunto de genes-alvo, segundo "modelos em cascata reguladora" feitos à imagem do conceito de cascata enzimática da Bioquímica: uma sequência de reacções em cadeia, em que uma reacção enzimática transforma uma proteína inactiva num enzima activo que por sua vez cataliza a activação de outro enzima e por aí adiante. Outros modelos em cascata são protagonizados por enzimas que realizam a transdução e amplificação intracelulares de um estímulo inicial recebido do meio extracelular — tudo caminhos regulatórios, às vezes combinados, que conduzem à indução, ou também à repressão (silenciamento), de um gene ou coorte de genes:

Cada locus tem uma região promotora (ou promotor) através da qual é regulada a sua transcrição. Por analogia com as origens de replicação, os promotores são segmentos do DNA que contêm sequências ("palavras-chave") especificamente reconhecidas por proteínas reguladoras, das quais depende a ligação da polimerase II do RNA. De facto, uma ligação efectiva do complexo enzimático de transcrição é o passo final de uma cascata de interacções envolvendo diversas proteínas e o promotor desse locus; algumas dessas interacções também desestabilizam os nucleossomas e desenrolam localmente a dupla cadeia para expor as purinas e pirimidinas para os emparelhamentos com os NTPs a incorporar na molécula de RNA.
O estudo de muitos promotores tem demonstrado sequências nucleotídicas envolvidas na regulação da transcrição, dispersas por vezes a longas distâncias a 5' do início da transcrição. De todos esses segmentos, dois são comuns a praticamente todos os promotores para a polimerase II do RNA, encontrando-se a uma distância (número de pares nucleotídicos) fixa do início (posição +1) da transcrição: são as boxes CCAAT e TATA, que nas posições –35 e –10 respectivamente constituem no DNA os pontos de contacto da polimerase II do RNA; as outras sequências que se conhecem definem o tipo celular, ou os estímulos, que podem actuar sobre aquele promotor, e isso comprova-se porque outros genes, até com funções muito distintas mas com padrões de expressão análogos, apresentam nos seus promotores algumas dessas sequências em comum. O repertório de sequências reguladoras da região promotora de cada gene há-de ser globalmente específico desse gene, mas parcelarmente comum com o de outros genes que são reconhecidos por proteínas reguladoras comuns. Isso permite por exemplo coordenar diferentes genes com um só estímulo dado à célula.
Dado que a transcrição só se faz sobre uma das duas cadeias do DNA, definiu-se uma nomenclatura que permite identificar a que cadeia diz respeito uma determinada sequência. O padrão é sempre a sequência presente no mRNA (e no transcrito primário), e atribui-se-lhe o símbolo + ; assim, o que a polimerase II do RNA faz no decurso da transcrição é ler na cadeia – do DNA e emparelhá-la com ribonucleótidos de modo a formar a sequência que lhe é complementar (+). Dado que as cadeias – em cada gene servem apenas de molde, quando se apresenta uma sequência de DNA mostra-se apenas a cadeia +, que é semelhante à informação (substituindo o T pelo U) presente no respectivo mRNA.
O transcrito primário não é ainda o mensageiro (o mRNA) a ser traduzido no citoplasma: as suas extremidades 5' e 3' (respectivamente, o primeiro e o último ribonucleótidos sintetizados) terão ainda que incorporar duas estruturas terminais protectoras da actividade das exonucleases do citoplasma: um nucleótido de 7-metilguanina invertido (cap), que protege a extremidade 5' deixando apenas exposto o seu 3'-OH, e uma cauda poliadenílica (poliA) na outra extremidade (os mRNAs das histonas constituem excepção neste segundo caso). E há, em quase todos os genes, uma série de segmentos intermédios do transcrito primário que são excisados ainda no núcleo por um processo a que se dá o nome de splicing. Resumindo, o transcrito primário é alvo de um processamento dentro do núcleo que consiste da adição de estruturas terminais e da remoção de segmentos intermédios. Este processo é esquematizado na figura 12:
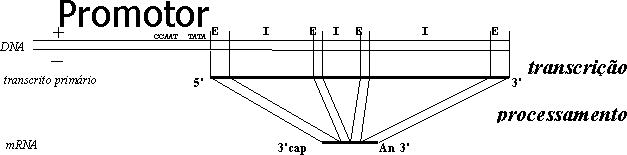
Figura 12 — Produção do mRNA após a transcrição e o processamento. Na cadeia + do DNA transcrito definem-se dois tipos de segmentos: os exões (E), cujas sequências transcritas persistem no mRNA, e os intrões (I), cujas sequências no transcrito primário são excisadas durante o processamento, deste modo não fazendo parte do mRNA.
O mRNA é translocado para o citoplasma através dos poros do envólucro nuclear, e uma vez no citoplasma, deverá ser traduzido pelos ribossomas, produzindo-se um polipéptido pela leitura dos codões que se sucedem ao codão de iniciação AUG no mesmo sentido da transcrição, isto é, no sentido 5' → 3' do mRNA. O cap é o ponto de ligação para a subunidade 40S do ribossoma, que faz a busca do primeiro trinucleótido AUG (cap....AUG.....3'; outros trinucleóticos podem ser os utilizados em certos mRNAs), onde se lhe junta a subunidade 60S para regenerar o ribossoma funcional; aí incia-se a síntese do polipéptido, pelo ciclo {identificação do codão → ligação do aminoacil-tRNA que emparelha correctamente → ligação peptídica → translação de 3 nucleótidos no sentido 3' → identificação do novo codão}, até surgir um codão de terminação, que sinaliza a dissociação do complexo ribossomal do mRNA e libertação do polipéptido sintetizado. Todos os mRNAs contêm codões de terminação (UAG, UAA ou UGA) necessários a definir a posição correcta do terminal carboxilo do polipéptido que codificam.
Todas as proteínas integrais da membrana celular, assim como as que são secretadas para o meio extracelular e as que se encontram na face externa da membrana celular — e ainda todo o conteúdo enzimático dos lisossomas — são traduzidas no retículo endoplásmico rugoso, donde são translocadas, através de vesículas do retículo (em diversos casos com maturação pós-tradução no complexo de Golgi) para os compartimentos de destino. Isso deve-se à presença, na extremidade amino dos respectivos polipéptidos ainda em polimerização, de uma sequência-padrão de resíduos de aminoácidos, chamada de péptido-sinal, assim designada por direccionar o complexo polirribossómico recém-formado para o retículo endoplásmico rugoso (codificada, por isso, numa grande diversidade de loci). Este péptido-sinal é excisado após "ancorar" os ribossomas ao retículo, enquanto a restante cadeia polipeptídica vai sendo sintetizada; no final da tradução, segundo os casos, o polipéptido é depositado no lúmen do retículo ou integrado na respectiva membrana.
Em contrapartida, muitos mRNAs não codificam o péptido-sinal e são por isso traduzidos no citosol, podendo as proteínas resultantes ou permanecer nesse compartimento, ou associarem-se às membranas (por exemplo a face citoplásmica da membrana celular), ou ainda serem translocadas para os plastos, mitocôndrias, e outros organitos. Com efeito, os genomas citoplásmicos codificam apenas uma parte limitada dos conteúdos proteicos dos respectivos compartimentos, havendo até complexos enzimáticos multiméricos (por exemplo das mitocôndrias) em que parte das subunidades são codificadas no organito, enquanto as restantes são codificadas no genoma nuclear, com tradução no citosol, e a translocação a partir deste é feita por proteínas especializadas conhecidas colectivamente como "chaperons".
A noção histórica de "1 gene, 1 enzima" está longe de ser geral. Nos procariotas muitos mRNAs são traduzidos em vários polipéptidos, porque o ribossoma pode iniciar a tradução a meio, bastando o reconhecimento da sequência 5'AGGAGGU3' (sequência de Shine-Dalgarno), que emparelha com o rRNA da subunidade pequena do ribossoma bacteriano; neste caso usa-se o termo operão para o segmento cromossómico que é transcrito, e respectivo promotor, e a cada segmento codificante dum polipéptido o termo cistrão. Os cistrões dum mesmo operão estão associados a uma mesma componente do metabolismo, sendo este um modo de terem a sua expressão coordenada.
Outras excepções, aliás bastante comuns, são certos vírus eucarióticos, que por outros mecanismos produzem mais do que um polipéptido por mRNA, mas de especial relevância é o splicing alternativo em genes eucarióticos, em que o mesmo transcrito primário pode produzir dois ou mais mRNAs diferentes, pela inclusão de diferentes séries de exões, assim diversificando o potencial funcional de cada gene. O estudo dos genomas eucarióticos em demonstrado que o splicing alternativo é mais a regra do que a excepção.
O DNA, apesar da sua estabilidade, é susceptível de sofrer mutações; a nível molecular elas classificam-se em mutações pontuais e rearranjos.
Mutações pontuaisA replicação e a transcrição não são livres de erros de emparelhamento, embora muitíssimo limitados. No primeiro caso, como esses erros são hereditários e amplificados pela transcrição e tradução, a sua ocorrência pode influenciar o fenótipo de várias maneiras, mas também pode não afectá-lo de todo. Quando se dá a substituição de um nucleótido por outro numa sequência, trata-se de uma mutação pontual (pontual porque afecta um par nucleotídico apenas). Se não se manifesta é uma mutação silenciosa ou sinónima, e de facto a grande parte das mutações pontuais que se detectam são silenciosas; mas se a mutação determinar um erro de reconhecimento de determinada região do DNA (nomeadamente mutações nos promotores que determinam a perda de afinidade dos respectivos reguladores pelo DNA nessa região), ou um erro de tradução (mutações missense, nonsense, frameshift, v. adiante), ou ainda se afectar o normal processamento do transcrito primário (por exemplo pode resultar na permanência de um intrão no mRNA, ou inversamente a excisão de um exão), então é provável que tenha um efeito fenotípico, embora não se possa dizer a priori qual.
|
A possibilidade de introduzir experimentalmente mutações pontuais ("site-directed mutagenesis"), em genes isolados in vitro, permite verificar os efeitos de cada mutação, a nível bioquímico e mesmo do organismo, com a substituição do gene normal pelo gene mutado. Este tipo de manipulação tem sido feito em diversas espécies experimentais, incluindo plantas superiores. |
A tradução de um codão no respectivo aminoácido define-se pelo código genético, e permite dizer se a substuituição de um nucleótido em determinado codão resulta na substituição do aminoácido codificado nessa posição do mRNA. Este código reside primariamente no grupo de enzimas que estabelece sem ambiguidades a correspondência entre o anticodão (uma sequência de três nucleótidos específica de cada tipo de tRNA que irá emparelhar, no complexo ribossómico, com o respectivo codão dos mRNAs) e um determinado aminoácido: as aminoacil-tRNA sintetases. Existem algumas variantes do código genético, especialmente nas mitocôndrias de alguns organismos, mas também nos plastos (semelhante ao das bactérias), de tal maneira que o antigo conceito de código "universal" deu lugar ao de código "padrão".
Uma das características deste código é a redundância entre codões (diz-se que é um código degenerado) ao nível sobretudo do nucleótido na 3ª posição: para muitos aminoácidos a 3ª posição é totalmente indiferente (a letra N designa "qualquer dos 4 tipos de nucleótidos"), e para quase todos os restantes a 3ª posição só tem de ser R (purina) ou Y (pirimidina).
|
Uma consequência da (quase absoluta) universalidade do código genético e da sua degeneração é a possibilidade que dá a diferentes espécies de organismos de divergirem no seu conteúdo relativo em pares G::C e mesmo assim codificarem proteínas com estruturas primárias idênticas de uns para os outros. |
Classificam-se as mutações pontuais em duas categorias:
a) As mutações missense definem-se pela codificação de um aminoácido diferente do normal. Os efeitos fenotípicos são em princípio tanto mais drásticos quanto maior for a diferença na natureza química das cadeias laterais dos resíduos dos aminoácidos em causa (por exemplo a substituição de um resíduo polar por um apolar, ou a inversão da carga eléctrica do resíduo), mas também dependem altamente do papel que esse resíduo desempenha na estrutura e função da proteína em causa: mesmo as chamadas substituições conservadoras (entre resíduos quimicamente semelhantes, por exemplo leucina, L, e isoleucina, I), se incidirem numa posição "sensível" da proteína (nomeadamente no centro activo), podem resultar na inactivação da proteína, ou numa actividade anormal da mesma. Por exemplo, a mutação CUA → AUA é missense (leucina em isoleucina) conservadora; a mutação CAU → GAU é missense (histidina, H, em aspartato, D) e inverte a carga eléctrica; a mutação CUA → CCA é missense (leucina em prolina, P) e, apesar de manter o carácter neutro do resíduo, pode ter efeitos muito drásticos se ocorrer numa hélice α da proteína; a mutação UGU → UAU é missense (cisteína, C, em tirosina, Y) e também pode ser muito drástica se impedir a formação de uma ponte dissulfureto (de cisteínas) envolvendo essa posição no polipéptido.
b) Nas mutações nonsense, um determinado codão de resíduo de aminoácido é substituído por um codão de terminação (por exemplo na mutação UGG → UGA), resultando num polipéptido truncado, que também pode ter efeitos muito diversos segundo os casos. Certas mutações supressoras de nonsense localizam-se em tRNAs que impedem que a tradução termine porque emparelham com o codão de terminação da mutação nonsense, introduzindo um aminoácido, possivelmente em missense.
Nas mutações frameshift, houve a inserção ou a deleção de pares de nucleótidos (em número não múltiplo de 3), fazendo com que no mRNA respectivo o ribossoma passe a identificar codões desfasadamente a partir desse ponto (a 3' da mutação, portanto), produzindo uma cadeia polipeptídica que não só tem uma série de mutações missense como tem um número de aminoácidos diferente (geralmente menor) do que o normal. Por exemplo, se a terceira adenina na sequência de codões AAG-AUU-UCC-UCA-AUG-GAA-U fosse removida, o péptido correspondente seria ...KFPQWN... em vez do ...KISSME... normal.
|
Certas proteínas são redundantes entre si, de modo que a inactivação da proteína codificada num dado locus não chega a ter efeitos fenotípicos (cf. interacções entre loci"), enquanto o segundo locus não for alvo de análoga inactivação. Mas a redundância entre loci, uma das consequências da duplicação genética, também pode abrir a possibilidade de dois loci com função semelhante poderem, pela mutação pontual ou pelo rearranjo cromossómico, divergir entre si. Este mecanismo, com profundas implicações funcionais e evolutivas, está na origem das famílias de genes, séries de loci (ligados ou não) que pela homologia parcial das sequências nucleotídicas denotam uma ancestralidade comum, isto é, a partir de um único locus que foi duplicado em vários. |
Muitas vezes a indução experimental de mutações pode substituir a introgressão para mutar os genes. Os procedimentos mais comuns e bem-sucedidos na prática baseiam-se na exposição a radiações ionizantes, nomeadamente os raios X e os raios γ, e também na imersão de sementes em mutagénios químicos ou o pincelamento de gemas com esses mutagénios. Em qualquer dos casos visa-se provocar mutações aleatoriamente, a uma taxa mais elevada que a espontânea, e pela selecção aproveitar as novas características eventualmente de interesse agronómico, ornamental, etc..
RearranjosTodas as variações estruturais detectáveis a nível citogenético (cf. "cromossomas"), assim como variações do mesmo tipo só detectáveis a nível nucleotídico (nomeadamente inserções, deleções, inversões, duplicações) são rearranjos do DNA. Por exemplo a inserção ou deleção dum número substancial de pares nucleotídicos no interior de um exão, mesmo que não seja em frameshift, acarreta uma alteração importante da estrutura da proteína, podendo com isso produzir-se um alelo funcionalmente alterado (a repetição adicional de certos tripletos de nucleótidos, geralmente codões para a glutamina, constituindo inserções de aminoácidos na estrutura primária da proteína sem alterar a restante sequência, dão um exempo disso, pois tem-se revelado a causa, pelo menos na espécie humana, de uma série de anomalias genéticas).
No lado positivo, muitas das variações importantes dentro das chamadas famílias de genes devem-se ao efeito criativo, com grande significado evolutivo, de novas combinações entre exões, promotores, etc., ou até a duplicação de loci, todas resultantes de rearranjos.
Os vírus e os viróides são entidades subcelulares com replicação autónoma no interior das células que parasitam. A actividade dos vírus resulta em geral na lise das células infectadas, libertando grandes quantidades de formas infecciosas (viriões) que deverão propagar-se a outras células numa vizinhança mais ou menos próxima, afectando o tecido e eventualmente destruindo-o. Porém há vírus que, tendo no seu ciclo replicativo um estádio de DNA em dupla cadeia, podem em vez de multiplicar-se imediatamente na célula hospedeira integrar-se no genoma desta (o segmento de DNA integrado é o provírus). Nesse estado, podem ser transcritos e daí as células hospedeiras caracterizarem-se pela presença de produtos virais mas sem disseminarem a infecção (apenas se duplicando o provírus pela síntese normal de DNA na fase S do ciclo celular). Pelo processo de indução viral, restaura-se a actividade replicativa autónoma iniciando-se um foco de infecção com a lise das células hospedeiras.
Os vírus (assim como os viróides) podem atingir elevadas concentrações dentro do tecido que infectam, sendo facilmente purificados em laboratório. Historicamente, os vírus foram uma via muito bem sucedida para a investigação de muitos dos aspectos moleculares da expressão dos genes em células animais e dos restantes eucariotas: dado que em quase todos os casos têm um material genético muito restrito, os vírus têm de aproveitar-se do aparato enzimático da célula hospedeira para completarem os seus ciclos reprodutivos (por exemplo, todos os ciclos virais incluem um estádio mRNA que monopoliza, em maior ou menor grau, os ribossomas da célula). Assim, a observação da actividade viral, na sua faceta de manipulação dos mecanismos presentes na célula hospedeira, é da maior utilidade para o estudo desses mecanismos em si.
|
Actualmente, certos componentes virais, através da síntese de genes quiméricos por engenharia genética, são utilizados na investigação fina de mecanismos de expressão dos genes, como por exemplo o tempo de semi-vida de um determinado tipo de mRNA em determinado tipo de célula, ou a análise da expressão de certos genes reguladores durante o desenvolvimento embrionário. |
O tratamento de viroses requer o isolamento do agente e a caracterização dos processos que lhe permitem replicar-se e/ ou expressar-se. Dado que os problemas que causa assentam na capacidade de propagar-se a partir de uma célula infectada, o tratamento passa pela interrupção do ciclo de lise, através da inibição de algum dos processos nele envolvidos.
|
Esta ênfase no metabolismo viral levou a classificar os vírus segundo o material genético presente no virião: há vírus que contêm como material genético DNA (de cadeia simples (–) ou de cadeia dupla), ou RNA (de cadeia simples, + ou – , ou de cadeia dupla), e com este critério definem-se modelos de replicação e expressão que são peculiares a esses grupos. |
O problema é que, quanto menos codificação residir no cromossoma viral, menos específico é o seu metabolismo, tornando-se sumamente difícil travar a sua propagação sem afectar também o organismo hospedeiro. O caso dos viróides é extremo: são apenas constituídos por uma molécula de RNA circular que auto-cataliza a sua própria replicação — tudo o que lhes é necessário são os NTPs e o ambiente (redox, temperatura, força iónica) que encontram nas células.
|
É por causa do grupo álcool 2' presente nos resíduos de ribose das cadeias de RNA (aludida na legenda da figura 9b) que este tem actividade catalítica de hidrólise ou de condensação (os chamados ribozimas constituem uma tecnologia que procura tirar partido disso); esta catálise é por exemplo aproveitada para a excisão dos intrões do transcrito primário, com o auxílio de pequenos complexos nucleoproteicos do núcleo (snRNPs) que emparelham com os pontos correctos de excisão. |
Os retrovírus são uma classe especial de vírus animais e vegetais (e presumivelmente fúngicos) que contêm uma molécula de mRNA na altura da infecção mas não a replicam como outros vírus de cadeia + : utilizam-na como molde para a retrotranscrição ("reverse transcription"), pela qual resulta uma molécula de DNA de cadeia dupla. Esta molécula de DNA integra-se no genoma da célula hospedeira e é transcrita intensamente pela respectiva polimerase II do RNA, em virtude do forte promotor viral presente no provírus.
Alguns dos elementos presentes no genoma aparentados com os genes de retrotranscriptase são os retroposões, classe de elementos móveis que são autonomamente capazes de duplicarem-se, integrarem-se, excisarem-se, recombinarem-se com os cromossomas, etc.. São, por assim dizer, versões benignas de vírus, pois também têm autonomia replicativa mas segundo parece não chegam a propagar-se entre células, ficando por isso prisioneiros do genoma a que pertencem. Parte do DNA altamente repetitivo são retroposões. Tal como a retrotranscriptase, têm uma larga utilização nas abordagens experimentais em Biologia Molecular.